
研究DNA与蛋白质互作时,通常需要有目标DNA或者目标蛋白质。如果研究时,只有处理材料,需要找寻处理材料中影响材料表型变化的调控因子,可以进行ATAC-seq实验。


图.100-10w细胞投入量ATAC结果
细胞投入量从100个到10万个,均能得到建库结果,且结果的拟合度较高。
已知材料中研究蛋白质,需要探究该目的蛋白质靶向的DNA结合区域,可以使用ChIP-seq、CUT&Tag、DAP-seq、CUT&RUN进行研究得到结合的DNA序列后,再利用EMSA、双荧光素酶报告基因、酵母单杂交实验、DNase I足迹试验等。若是研究时,发现可能出现共调控的区域,还需要进行CoIP或者GST pull down实验进行验证。
ChIP-seq、CUT&Tag、DAP-seq、CUT&RUN介绍
ChIP
ChIP是研究DNA与蛋白互作的传统经典方法,需要将样本进行甲醛交联后再用超声设备进行片段化处理,之后利用特异性抗体进行靶向富集,将富集得到的DNA进行建库测序分析即得到靶向蛋白质互作的DNA信息。
CUT&Tag是研究DNA与蛋白互作的新型技术,利用转座酶对抗体靶向区域进行切割,将切割得到的DNA序列可以一步扩增得到DNA文库。相对于ChIP-seq实验,CUT&tag具有可重复性强、背景噪音低、起始量要求低等优点。
Multi-CUT&Tag是利用新兴的纳米抗体偶联Tn5(Nanobody-Tn5),利用纳米抗体识别一抗,使得Tn5被纳米抗体靶向到特定位置发挥靶向切割,将切割得到的DNA序列进一步扩增得到DNA文库。相对于传统CUT&Tag技术,Multi-CUT&Tag可以实现一份样本,同时研究两种靶标,节约样本和时间,实验无需二抗,减少实验流程。
CUT&RUN是研究DNA与蛋白质互作的新型技术,利用Mnase酶对染色质开放区域进行切割,之后将切割得到的DNA进行建库以获得靶向蛋白质互作的DNA信息。
DAP-seq是研究DNA与蛋白互作的技术,它属于体外ChIP-seq,主要针对一些非模式物种构建转基因体系或者过表达实验不容易操作的样本,本实验的优点是无需购买抗体即可实验。
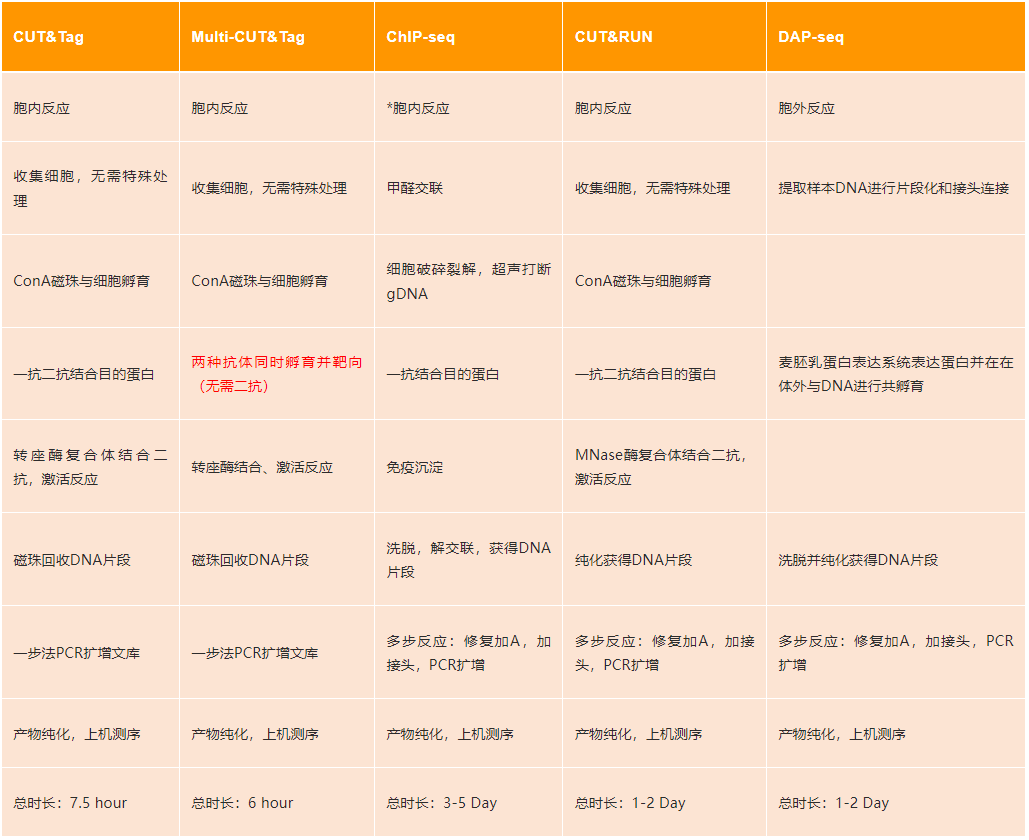


图.不同组蛋白修饰的CUT&Tag及Multi-CUT&Tag实验结果
Multi-CUT&Tag可以实现一份样本,两种不同组蛋白修饰的研究。组蛋白修饰同时研究的数据拆分后的结果与常规单靶标研究结果一致。
