大家好,我是伯小远的哥哥伯大远。今天由伯大远来跟大家唠唠RNA中的siRNA、shRNA和miRNA。2000年,《科学》杂志将“重识RNA”选为“十大科技突破”之一,如果说RNA是生物科学研究领域的“明星”,那siRNA、shRNA和miRNA就是“明星”中的“巨星”。
说起siRNA、shRNA和miRNA就不得不提RNA干扰(RNA interference,RNAi),它是指由一段短双链RNA引起的基因沉默现象。1998年,Andrew Z. Fire等在实验中发现,单独使用纯化的正义RNA链或者反义RNA,不能导致秀丽隐杆线虫的肌肉抽搐,作为对照加入的双链RNA则具有极强的抽搐效果[1]。加入的双链RNA的抑制效果远强于理论上1:1配对时的反义RNA的抑制效果,这种现象被命名为RNAi(想了解更多RNAi的发现过程,可以阅读王秀杰研究员的《RNA干扰:双链RNA引起的基因沉默机制-2006年诺贝尔生理学或医学奖成果简介》)。

2001年“RNA干扰”作为当年最重要的科学研究成果之一,入选《科学》杂志“十大科技突破”;2002年,《科学》杂志将“Small RNA & RNAi”评为2002年度最耀眼的明星。同时,《自然》杂志亦将Small RNA评为年度重大科技成功之一。2003年,小核糖核酸的研究再次入选《科学》“十大科技突破”。2006年,Andrew Z. Fire与Craig C. Mello由于在RNAi机制研究中的贡献而获得诺贝尔生理及医学奖。RNAi现象发现8年后就获得诺贝尔奖,这在诺贝尔生理及医学奖历史上是罕见的。2018年,基于RNAi的基因沉默药物上市,又双叒叕被《科学》评为“十大科技突破”。可以看出,关于RNAi的研究是多么热门而重要,而RNAi研究的主角就是本文要介绍的siRNA、shRNA和miRNA。
图1.siRNA的结构

图2.shRNA的结构
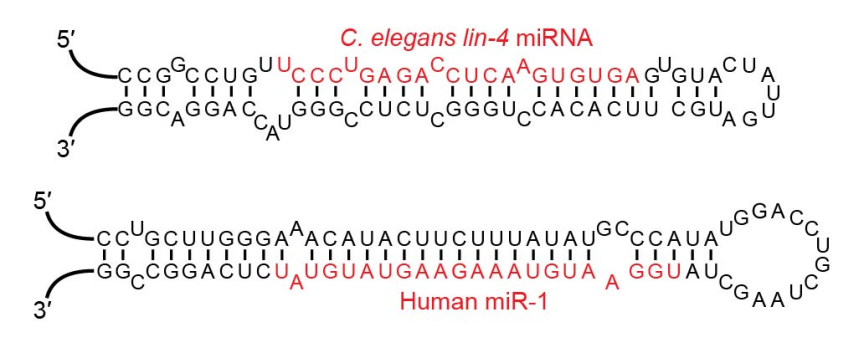
图3.miRNA的结构

1、三者的成熟体都是长度在20-25nt的小RNA,siRNA是3’端有两个游离的碱基,5’端有磷酸基团的双链RNA,shRNA经过加工形成siRNA,miRNA则最终形成单链RNA。
2、siRNA一般是人工合成的线性双链RNA(dsRNA),shRNA、miRNA则是通过发卡结构形成的局部双链RNA。siRNA和shRNA的双链区是完全互补配对的,而miRNA绝大多数情况下是不完全互补的。
3、从结构上看shRNA和miRNA更相似,事实上shRNA在功能上与siRNA更接近。shRNA在细胞内被Dicer酶切割后形成siRNA,通过siRNA途径行使干扰功能,而miRNA则是通过另一条不同的途径调控目标基因。
他们之间有很多相似之处,特殊情况下甚至可以相互替代,却在RNAi现象中各自发挥着不同的作用,他们之间到底有什么关联和差异,下面伯大远就跟大家详细介绍一番。

shRNA实际上是通过载体构建在体内获取siRNA的一种方式,两者结构本质上是相同的,但在功能上有所差异。同属于小分子RNA的miRNA与siRNA和RNAi有着莫大的关联,但是miRNA和siRNA又有着多方面的差异。下面就分别从shRNA和siRNA以及miRNA和siRNA介绍他们的联系和区别。
shRNA和siRNA
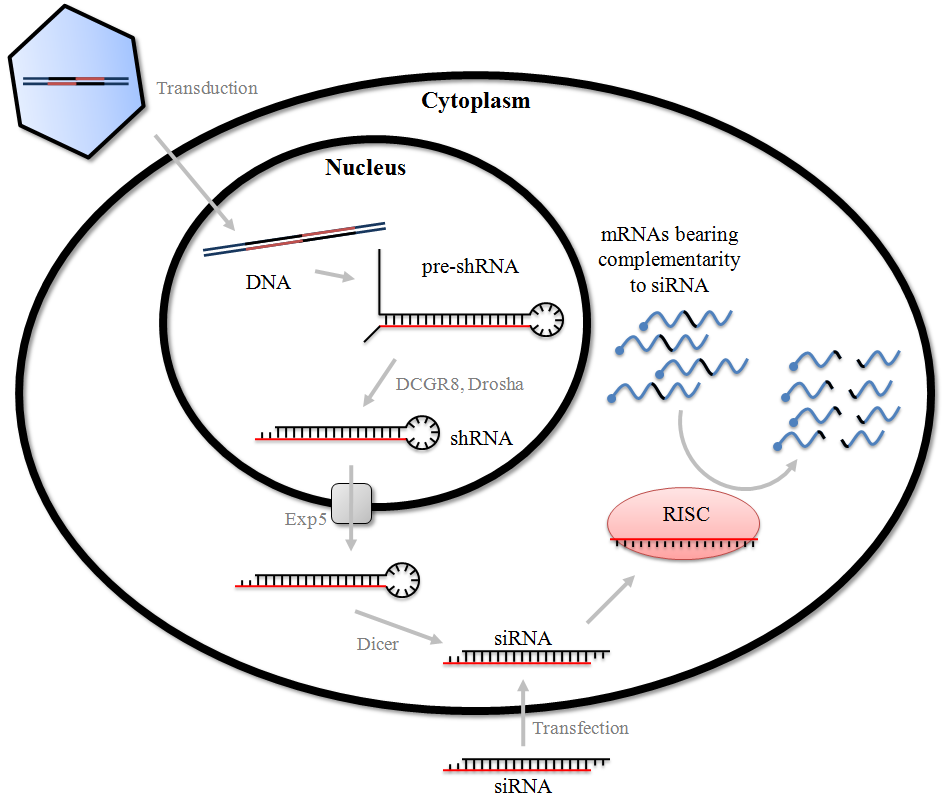
图4.shRNA与siRNA调控基因的原理图
上图是shRNA和siRNA在细胞内进行基因调控的基本原理图,可以看出两者是殊途同归的。siRNA可以在体外合成,但也常用较长的双链RNA作前体,dsRNA通过转染进入细胞,在细胞质内经过一系列酶处理,形成成熟siRNA,然后与RISC(RNA-induced silencing complexes)结合,最后与靶标mRNA配对,导致靶标mRNA的降解。而shRNA需要依托于载体、病毒、农杆菌等媒介,通过转染/侵染进入细胞,在细胞核中转录形成茎环双链,经过Drosha等酶的加工,形成成熟shRNA,然后转运至在胞质,在核糖核酸酶的作用下形成siRNA,进入RNAi路径。
既然shRNA和siRNA是殊途同归的,两者可以等同吗?当然不行咯,有以下几个原因:
1、 转染途径
2、干扰途径不同
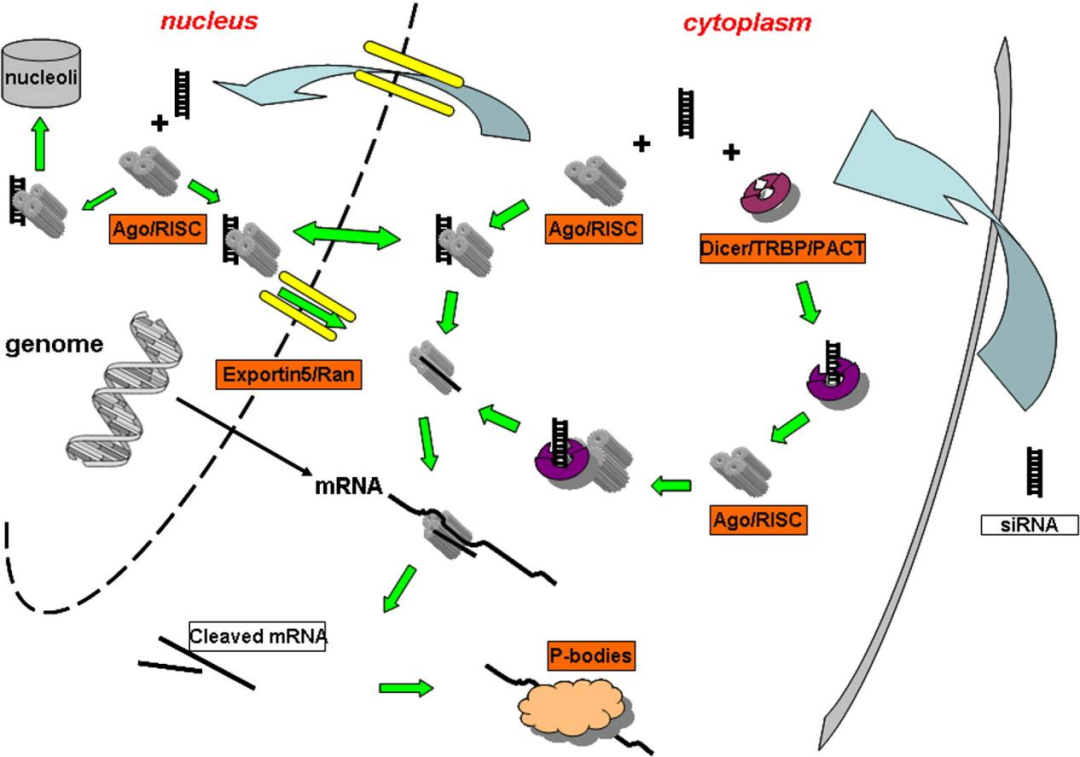
图5.siRNA干扰途径
据Jarve等[2]的报道,长双链RNA在转染细胞后15min内转移到细胞核中,然后在接下来的4小时内以完整和解离的形式散布到细胞质中。在胞质中,长双链RNA与Dicer/TRBP/PACT(TRBP是一种双链RNA结合蛋白,不同物种的名称不同,果蝇中是R2D2;PACT是一种激活蛋白)形成复合物,Dicer的双链特异性的核糖核酸酶III活性能够将长双链RNA处理成带有两个游离碱基的长度为21-23nt的siRNA。随后这些siRNA片段与RISC结合,如果转入细胞的是经过完美设计和加工的siRNA,可以绕过Dicer/TRBP/PACT的加工直接与RISC结合,RISC由Argonaute-2、Dicer和TRBP蛋白组成。再然后siRNA的两条链分开,其中一条链从复合物上离开,携带反义链的RISC复合物识别靶标mRNA,最终完成mRNA的切割与降解。
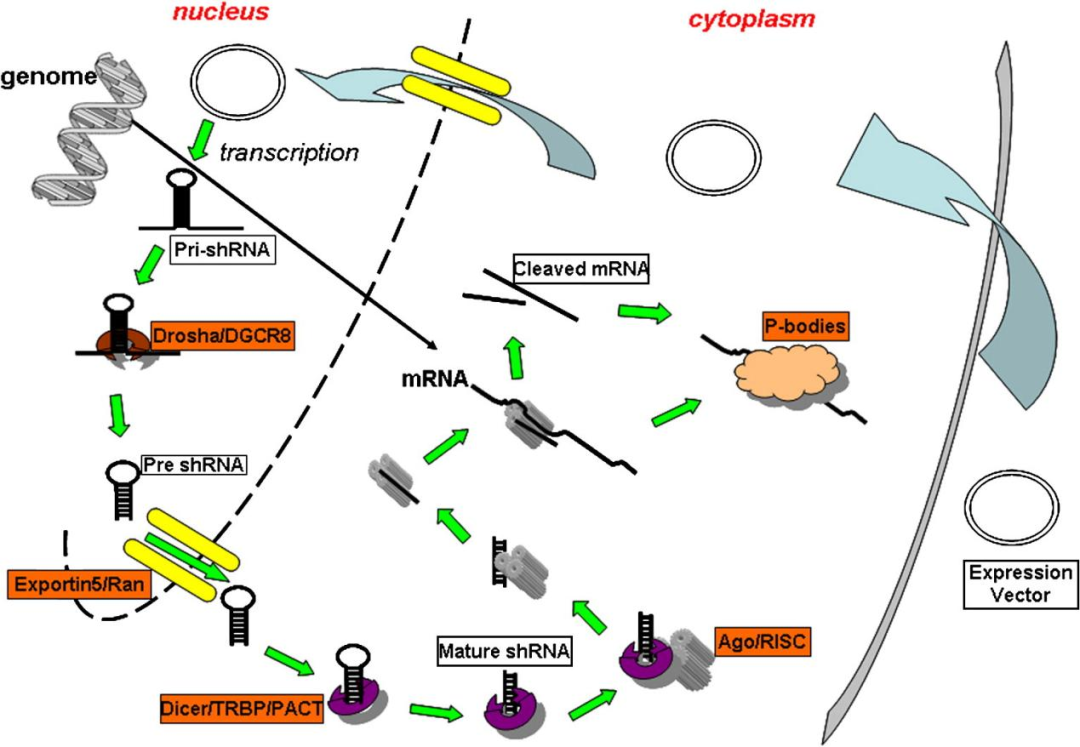
图6.shRNA干扰途径
与siRNA不同,shRNA在细胞核中合成。shRNA载体经瞬转或稳转进入细胞后,首先运输到细胞核,在细胞核表达后被Drosha/DGCR8复合酶加工形成pre-shRNA,然后由Exportin-5蛋白转运到细胞质中,在细胞质Dicer复合酶作用下去除环状序列形成siRNA。然后与RISC结合并离去其中一条RNA链,复合物识别mRNA,导致其降解。可见shRNA出核后与dsRNA的加工途径基本一致,最终也是形成siRNA的结构,但其稳定性和可持续性更强。想更详细的了解shRNA与siRNA的干扰途径差异,可以参考Donald等[3]的综述。
3、作用时间
4、表达水平
5、脱靶概率
miRNA和siRNA
如果按照RNAi的早期定义,miRNA是不属于该范畴的。虽然1993年就发现了miRNA,但其明确的功能研究稍晚于siRNA,而RNAi的定义主要是针对siRNA的。2001年,Science连续三篇文章报道了秀丽隐杆线虫miRNA的特点和功能,揭开了miRNA在生物生长发育过程中的重要调控作用[7-9]。随着研究的深入,已经发现miRNA与siRNA有很多相似之处,一些情况下miRNA与siRNA甚至使用相同的基因干扰途径。那么,miRNA介导的基因调控到底属不属于RNAi?伯大远没有找到明确的文献,但很多文献中将miRNA视为RNAi的另一种途径,本文支持这种观点。即siRNA、shRNA、miRNA分别是RNAi的不同途径,他们之间是相互补充的。
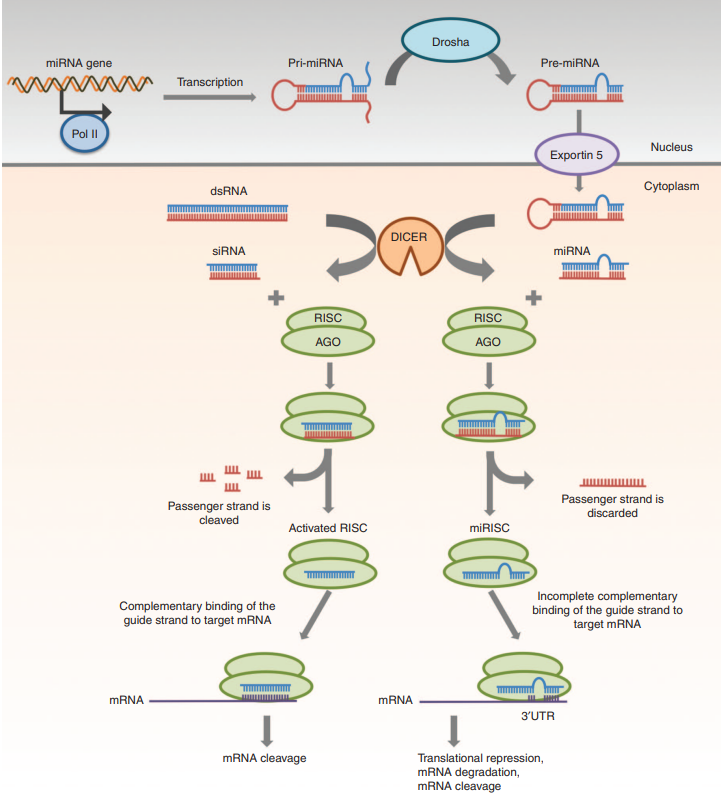
图7.miRNA与siRNA的RNAi途径
上图总结了miRNA、siRNA在细胞内执行RNAi的途径,前面已经介绍了siRNA的途径,很容易发现miRNA途径与shRNA途径高度相似,miRNA载体在细胞核中转录后经 Drosha酶处理形成pre-miRNA,pre-miRNA经Exportin5等蛋白运输出核,在细胞质经Dicer等酶处理去除茎环结构,随后装载到RISC上离去另一条RNA链,miRNA-RISC与靶标mRNA以非完全互补的方式配对,最后或导致mRNA降解或抑制mRNA的翻译,来干扰靶标基因的表达[7]。
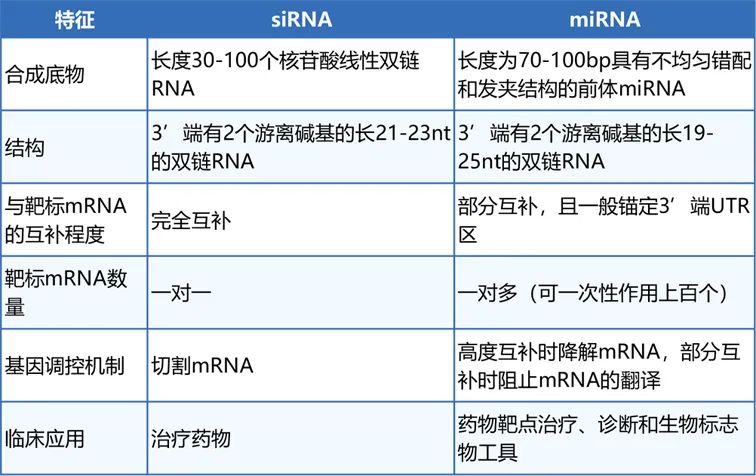
miRNA和siRNA很容易混淆,他们有许多共同点也有许多不同点。为了能够清楚地让大家弄清两者之间的差异之处,伯大远将从来源、加工途径、干扰功能三个方面来说明。
1、来源
siRNA通常是外源的,如病毒感染和人工插入的dsRNA被剪切后产生外源基因进入细胞,其作用瞬时的、不可被遗传的;而miRNA是内源性的,由非编码的pri-mRNA经过剪切加工而成,是生物体长期进化过程中自己形成的一套调控系统,在生物体中的表达具有时序性、保守性和组织特异性。
2、加工成熟
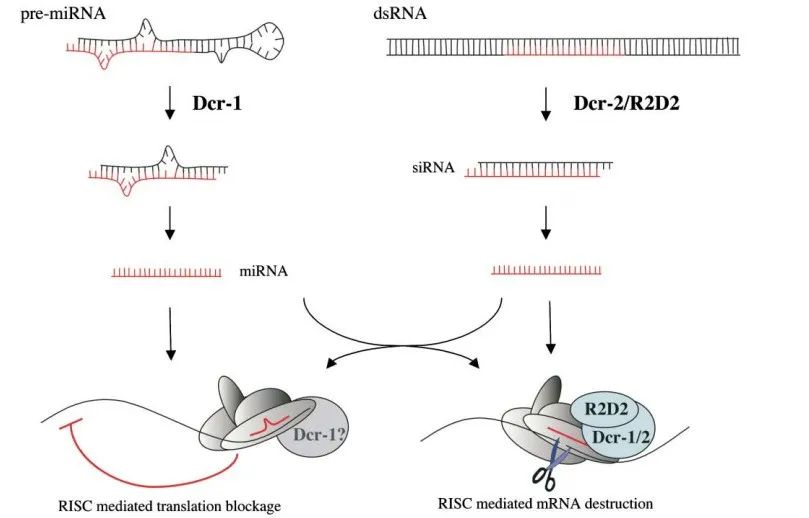
图8.果蝇中的RNAi模型
siRNA的直接来源是长双链RNA,经过Dicer酶切割形成双链siRNA,而且每个长双链RNA能够被切割成不定数量的siRNA片段。而miRNA需要在细胞核中转录剪切后释放到胞质,Dicer酶切割成熟。在人、秀丽隐杆线虫中Dicer酶只有一种,因此siRNA和被相同的Dicer酶成熟,而果蝇、拟南芥分别由2、4个Dicer酶,siRNA则是被不同的Dicer加工,在果蝇中pre-miRNA被Dicer-1及其伴侣蛋白Loqs加工,而dsRNA则被Dicer-2和R2D2加工。
3、干扰功能
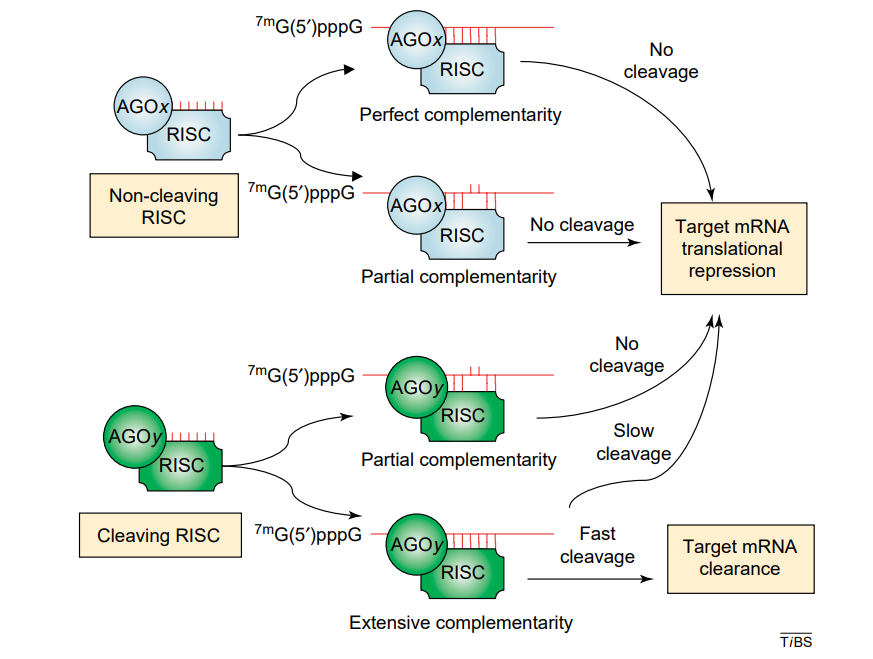
图9.miRNA与siRNA的干扰机理
成熟的siRNA和(双链状态的)miRNA分别被装载到RISC上,离去一条链后,携带反义RNA的RISC结合到mRNA上,分别通过mRNA降解和翻译抑制途径抑制靶标基因的表达。小RNA种类不同,两种RISC也是不同的。RISC的核心酶是AGO蛋白家族的Argonaute酶,它们在大多数物种中有多个成员,在果蝇和人细胞中,miRNA与AGO-1蛋白结合产生非切割RISC,通过抑制mRNA翻译干扰靶标基因;siRNA与AGO-2蛋白结合产生切割RISC,通过切割和降解mRNA干扰靶标基因(下图)。在拟南芥中,主要由AGO-1参与siRNA或完全互补的miRNA导致的mRNA降解。RISC的组装和靶向mRNA的机理非常复杂,不同物种间也有很大差异,想要了解更多可以参考Guiliang等[9]的报道。
此外,有些同学可能会疑惑:miRNA通常定位于靶标基因的3’-UTR区,且不会切割mRNA,而mRNA的翻译是从5’端开始的,miRNA是如果抑制翻译的呢?实际上,miRISC结合到mRNA的3’-UTR区后,仍然可以通过空间折叠影响到5’区,比如竞争翻译起始因子与mRNA的结合,诱导mRNA的去甲腺苷酸化,从而抑制mRNA的环化,诱导核糖体过早脱落等。这里不再赘述,感兴趣的可以参考Richard等[10]的综述。

到底是选择miRNA、siRNA,还是shRNA?
前面已经介绍了三者之间的差别,相信大家可以根据实验目的,判断出选择哪种干扰途径最合适。伯大远把一些建议汇总如下:
1、细胞便于转染或电穿孔转化,研究者关注周期,想快速获得结果,或者想通过荧光分子或其他分子标记检测小RNA,首选siRNA,直接由RNA合成公司合成,推荐选择PAGE及以上的纯化方式;
2、想彻底沉默某一基因且想特异性的沉默某基因,或者想通过诱导在特定时期产生干扰效应,或者想沉默长半衰期的蛋白,或者想获取能稳定遗传的细胞,首选shRNA;
3、植物基因沉默,首选shRNA;
4、基于RNAi的生物性状研究,首选shRNA;
5、内源非编码小RNA调控研究,首选miRNA;
6、想研究一种小RNA对某性状所有调控路径的影响,首选miRNA;
7、靶标基因不能够被完全沉默,否则会导致细胞死亡,首选miRNA;
8、咨询伯小远or伯大远吧

怎么设计呢?
siRNA

表中的大多数网址已经不可用了,其中http://sirna.wi.mit.edu/home.php(需要简单注册,但功能强大,给出了siRNA的热力学差异,界面也比较友好,强烈推荐),https://sfold.wadsworth.org/cgi-bin/sirna.pl(无需注册,靶标序列要求在250bp以内,不够简洁但内容丰富),http://sidirect2.rnai.jp/(siDirect 2.0,无需注册,界面也比较友好,比较推荐),https://www.invivogen.com/sirnawizard/(invivogen公司的在线工具,提供siRNA靶标搜索、加扰对照,提供对应的shRNA设计)[11]。
shRNA

shRNA在体内通常依靠RNA聚合酶III进行转录,常用人U6和人H1启动子驱动,据Petri等[12]的报道,在小鼠大脑中U6启动子驱动shRNA的干扰效果比H1启动子更好,因此本文推荐优先选择U6启动子。
shRNA的设计要点如下:
1. 选择靶标:参照siRNA。
2. 由于polyT结构是RNA聚合酶III的终止信号,shRNA序列中一定要避开4个及以上连续的T。
3. 使用U6或7S K启动子时,请选择以G开头的靶序列。
4. 避开与单核苷酸多态性(SNP,http://www.ncbi.nlm.nih.gov/projects/SNP/)区域重叠的靶标位点[13]。
5. 对照设置除了加扰和非靶向对照,还应该有个空载对照。
6. 使用在线工具:
https://rnaidesigner.thermofisher.com/rnaiexpress/sort.do(Thermo公司提供的RNAi设计工具,无需注册,提供siRNA、miRNA、shRNA设计,能进行一些动物的特异性搜索),https://biosettia.com/support/shrna-designer/(无需注册,简便但功能弱小,无特异性搜索)。
miRNA
①miRNA在结构上高度类似shRNA,其设计原则可以参考shRNA,但有两点不同需要注意:
a. miRNA是内源的,因此不再涉及靶标设计,而是从已知的miRNA库中选择,或者从文献中选择,比如已知同种属A物种miRNA对A基因的调控,想研究B物种中该miRNA是否能调控A基因的同源基因。早期研究认为miRNA主要靶向mRNA的3’-UTR区,虽然现在发现越来越多靶向CDS区和5’-UTR的miRNA,但选择时仍然推荐优先考虑3’-UTR区,尤其是动物细胞,植物细胞可以考虑CDS区和3’-UTR区。
b. pre-miRNA有一定的框架模式,A物种的pre-miRNA框架,不一定能在B物种中正确加工成熟,因此跨物种研究时,推荐试用目标基因宿主的pre-miRNA框架。
②也有直接在U6启动子后面接成熟miRNA序列的例子,但不多见也不推荐。
③miRNA表达也大多用U6和H1启动子,他们都是组成型启动子,shRNA和miRNA途径有一个最大的优点,是可以使用诱导型启动子,在特定时期或组织研究RNAi现象。
④miRNA设计也有一下参考数据库:

怎么分析干扰结果?
转录层面
①RT-qPCR分析
②RNA-Seq分析
蛋白层面
①双荧光素酶法分析
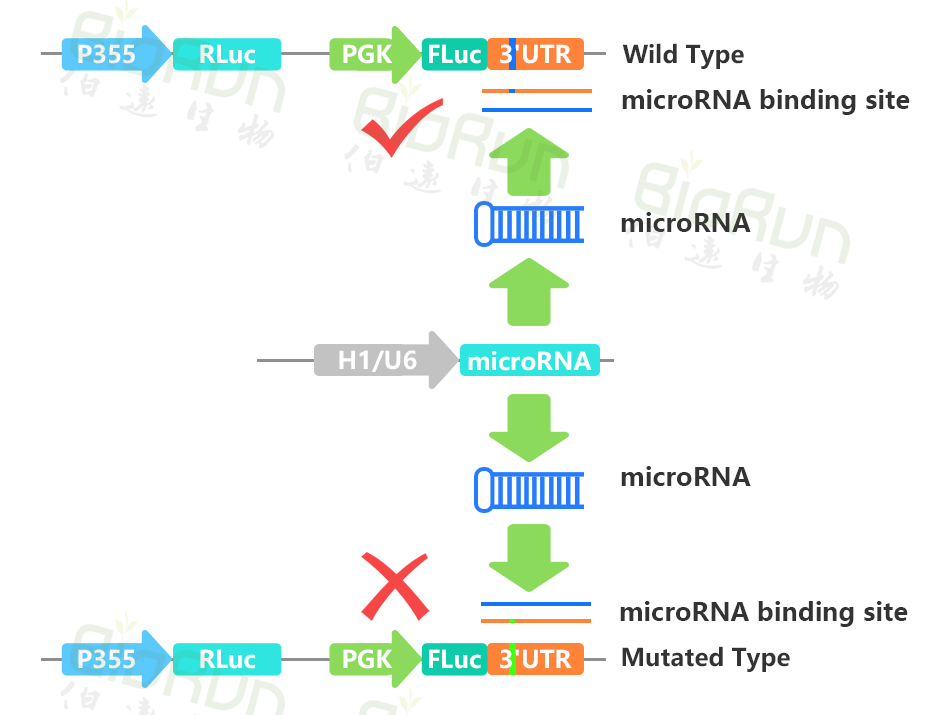
②WB分析
好了,到这里关于3个小RNA在RNAi中的异同点就讲完了,不知道你看完之后有没有豁然开朗的感觉呢!作为伯小远的哥哥今天首次登场,真的很用心的再为大家总结这方面的知识哦!从它们发挥功能的机制到设计引物以及最后的检测都进行了归纳总结,相信很多小伙伴在之前的研究中对这几个小RNA总是存在一些疑惑,既然这样,那就收藏这篇干货吧,让科研从此不再有困惑!
